2-6節
中国論文は捏造である
仕切り直し
ハンブルグ論文がリードデータの長さを気にしていたので、当研究所でもリードデータの長さについて少し議論してきましたが、結論としてはそこには大して意味がないだろうという考えに至りました。
そこでハンブルグ論文が指摘していた通り、リードデータをアセンブルすると論文と異なる結果が得られるのかについて確認したいと思います。論文でも述べられていましたが、論文と同じバージョンのアセンブラは入手できなかったので、ここでは最新のバージョンのアセンブラを使用しています。
- Megahit(Ver.1.2.9)
- Trinity(Ver.2.15.0)
アセンブル結果
アセンブルした結果は以下の通りです。
| アセンブラ | コンティグ数 | 最短コンティグ | 最長コンティグ |
| Megahit | 29,369本 | 200塩基 | 29,809塩基 |
| Trinity | 150,029本 | 184塩基 | 29,879塩基 |
ハンブルグ論文とほぼ変わらない結果が得られました。再掲します。
| アセンブラ | コンティグ数 | 最短コンティグ | 最長コンティグ |
| Megahit | 28,459本 | 200塩基 | 29,802塩基 |
| Trinity | 157,283本 | 201塩基 | 29,875塩基 |
中国論文とも比較してみます。
| アセンブラ | コンティグ数 | 最短コンティグ | 最長コンティグ |
| Megahit | 384,096本 | 200塩基 | 30,474塩基 |
| Trinity | 1,329,960本 | 201塩基 | 11,760塩基 |
ハンブルグ論文が主張していたように、論文と異なる結果が確認できました。
塩基配列の確認
次に塩基配列がどうなっているのか確認してみます。まずMegahitの最長コンティグと新型コロナウイルスの塩基配列(MN908947.3)と比較してみます。
比較には、「ApE」というソフトを使いました。
下の表は、比較した結果です。マッチ数というのは一致した塩基数です。ミスマッチ数とは一致しない塩基数です。ギャップ数とはお互いの塩基長が異なるが故に不足している塩基数を意味しています。
| アセンブラ | マッチ数 | ミスマッチ数 | ギャップ数 |
| Megahit | 29,808塩基 | 0塩基 | 96塩基 |
| Trinity | 29,874塩基 | 0塩基 | 34塩基 |
両者のアセンブラでもほぼ完全に再現していることが分かります。ここで問題になるのは新型コロナウイルスの塩基配列はMegahitの最長コンティグに対してアライメントを行って得られたものであるということです。ですから、アライメントした塩基配列を再現していることになります。そのため、アライメントされる前の塩基配列、すなわち論文で得られた最長コンティグを確認することができません。
塩基配列一致の構造
では、もう少し塩基配列がどのように一致しているのか確認したいと思います。
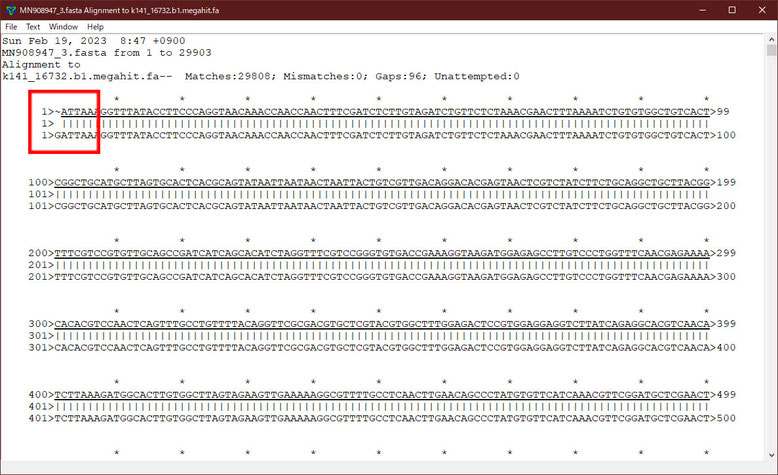
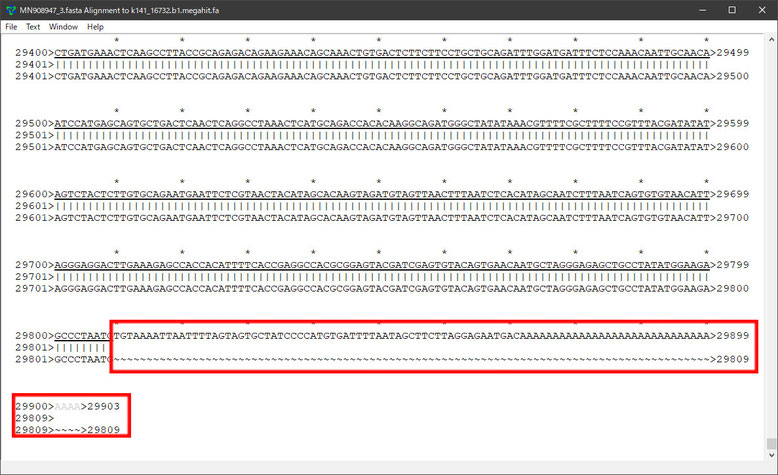
下の画像はMegahitで得られた最長コンティグと新型コロナウイルスの塩基配列(MN908947.3)を比較したものです。画像から分かるように、先頭に1塩基分のギャップがあり、後尾に95塩基分のギャップがあります。ギャップとギャップの間は完全に再現しています。
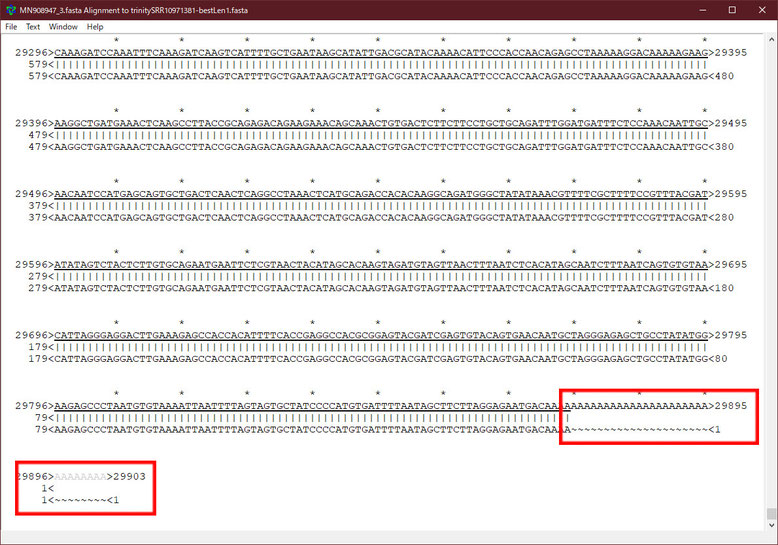
次の画像はTrinityの最長コンティグとMN908947.3を比較したものです。この場合もMegahitと同様に先頭と後尾にギャップがあります。先頭は5塩基分。後尾は29塩基分です。それ以外は完全に再現しています。ただ面白いことに、Trinityは向きが反対に再現しています。

中国論文は捏造である
以上のように、当研究所でもハンブルク論文とほぼ同じ結果が得られました。明らかに中国論文の結果と全く異なります。アセンブルした結果、アライメントした後の遺伝子配列が得られるとは考えられません。しかも、Megahitの最長コンティグにアライメントした結果が、Trinityでも再現できるというのはかなり奇妙です。何故なら中国論文ではTrinityの最長コンティグはMegahitの最長コンティグの半分も満たなかったからです。
公開されいてるリードデータ(SRR10971381)は、MN908947.3に行ったカバレッジが中国論文とほぼ同じであるため、中国論文で使用されていることは間違いありません。ですが、MN908947.3を決定する以前のリードデータと決定した後のリードデータは異なります。MN908947.3を再現できるように加工してあると思われます。リードデータは電子データですから、そんなことは難しいことではありません。
仮想化技術はリードデータにある
以上のことから、一つの仮説が思い浮かびます。武漢以外の研究所でMN908947.3を再現できるのは、リードデータを加工しているからではないか?ということです。つまり、適当な検体を手に入れて次世代シーケンサーにかけてリードデータを得る。その後、得られたリードデータに対して決められた手順で加工しているのではないかと思われます。
MN908947.3を再現するためには次の条件が必要だと考えます。
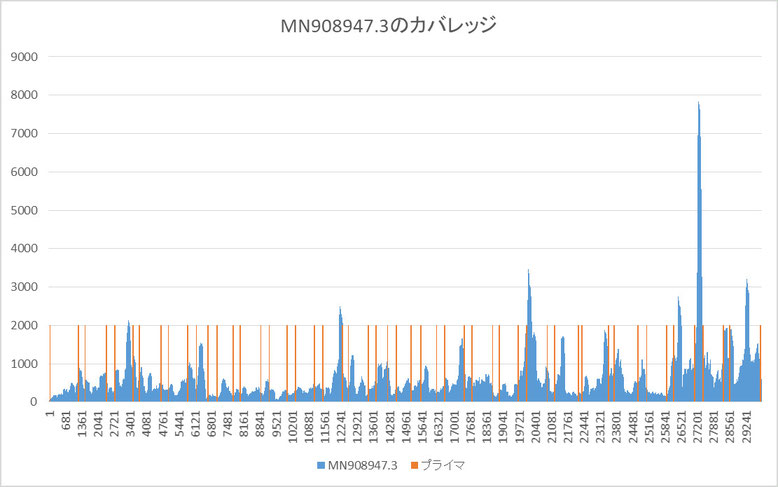
- MN908947.3の全域にわたってカバレッジが覆っている。
- 複数の組み合わせを作るようなリードが存在しない。
1番の条件は当然だとして、2番の条件ですが、このようなリードが存在すると途中から枝分かれして、別の遺伝子配列を作る可能性が出てくるので、再現できなくなります。ですから、そのようなリードを排除するような加工をしているのではないでしょうか。
また1番の条件を満たすために、26本のプライマーが意味を持ってくるように思います。このプライマーを用いて検体中の遺伝子物質の中でMN908947.3を覆う遺伝子物質を増幅させて、全体をカバーしやすくしているのかもしれません。だから、プライマー付近のカバレッジが異様に高い値を示しているのかと思われます。