2-5節
リードの長さの意味
この章で紹介している論文をハンブルグ論文と呼ぶことにします。
この節ではハンブルグ論文の内容を再実験して確認し、当研究所の仮説を検証したいと思います。
使用した環境及びソフト
今回の作業を行った環境は以下の通りです。
- Windows10
- WSL2
- Ubuntu(20.04)
使用したソフトは以下の通りです。
- fastp
- bowtie2-build(2.4.5)
- boutie2(2.4.5)
- samtools(1.16)
公開リードデータ
NCBIからダウンロードした公開リードデータ(SRR10971381)をfastpで不良データを排除しました。
| ペアエンドリードデータ | 本数 | 最短長 | 平均 | 最長長 |
| SRR10971381_1.fastq(順方向) | 14,328,298本 | 31塩基 | 137.8塩基 | 151塩基 |
| SRR10971381_2.fastq(逆方向) | 14,328,298本 | 31塩基 | 137.4塩基 | 151塩基 |
| 合計 | 28,656,596本 | 31塩基 | 137.6塩基 | 151塩基 |
このペアエンドリードのリード長の分布をグラフにしたものを以下に示します。
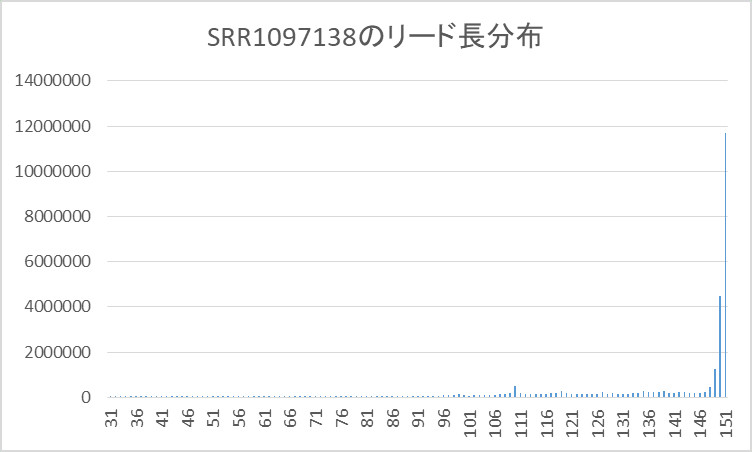
150塩基以下の分布が分かりにくいのでレンジを変えたグラフを以下に示します。

このグラフから分かるように151塩基長のリードがほとんどで、全体の約41%を示しています。
ハンブルグ論文では、150塩基長のリードが新コロゲノムの構造を決めていると予想していましたが、中国論文を読みなおすと、150塩基長のリードを用意していることが分かりました。
ですから、150塩基長のリードが大半を占めているのは、意図的であるということです。
つまり、150塩基長のリードが新コロゲノムの構造を決めているという予想は間違いであることが分かりました。
MN908947.3のカバレッジ
上記リードデータをbowtie2-buildとbowtie2を用いてカバレッジを求めました。
そのカバレッジと中国論文で設定したプライマ(52個)の位置をグラフにしたものを下に示します。
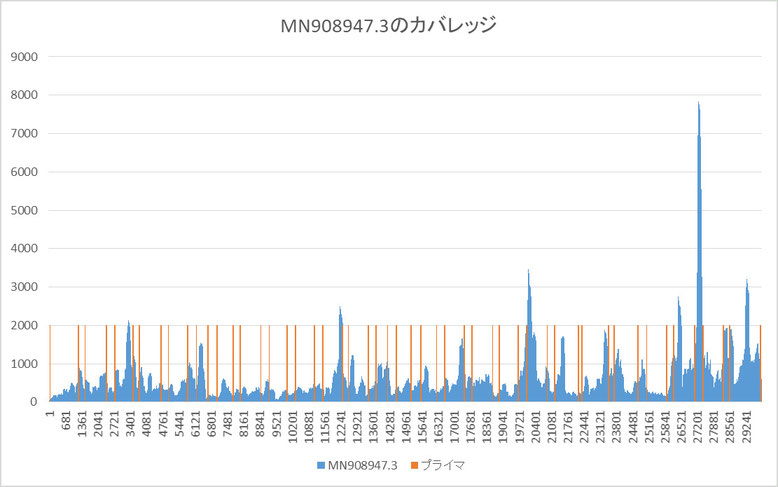
MN908947.3にマッチしたリードは以下のようになりました。
| ペアエンドリードデータ | 本数 | 最短長 | 平均 | 最長長 |
| SRR10971381_1.fastq(順方向) | 59,033本 | 31塩基 | 145.9塩基 | 151塩基 |
| SRR10971381_2.fastq(逆方向) | 59,033本 | 31塩基 | 145.9塩基 | 151塩基 |
| 合計 | 118,066本 | 31塩基 | 145.9塩基 | 151塩基 |
次にMN908947.3にマッチしたリードの長さの分布を見てみます。

ほとんどが151塩基のリードであることが分かります。
肝心の100塩基以下の分布が見えないので、レンジを変えたグラフを示します。

ハンブルグ論文と異なる結果が出ました。
比較のために再掲します。

全然違います。
理由は定かではありませんが、ハンブルグ論文の結果から私が立てた仮説は成り立たないことが分かりました。
繰り返しになりますが、150塩基のリードが新コロゲノムの構造を決めているという仮説は間違っていました。
リードにヒトゲノムが含まれているか
ここで一番検証したいのは、武漢以外で新コロゲノムを再現することができる理由として、リードにヒトゲノムやバクテリアゲノムが含まれているという仮説です。
プライマ付近でスパイク状にカバレッジが偏っている現象は、特異的なプライマを使って、新コロゲノムを再現することのできるリードを大量にコピーしているのではないかと言う仮説です。
これを検証するために、プライマにマッチするヒトゲノムやバクテリアゲノムがあるのか調べます。
まず、中国論文に掲載されているプライマデータを使って、下に示すfastaファイルを作りました。
>WHCV-F1
CCAGGTAACAAACCAACCAACTT
>WHCV-R1 Reverse Complement
GTGTGTTCTCTTATGTTGGTTGCC
>WHCV-F2
CAACCAAATGTGCCTTTCAACTC
>WHCV-R2 Reverse Complement
AGGTTACTTTTGGTGATGACACTGTG
>WHCV-F3
TGTCACGCACTCAAAGGGATT
>WHCV-R3 Reverse Complement
TTCGCACAAATGTCTACTTAGCTGTC
>WHCV-F4
ATGCCATGCAAGTTGAATCTGAT
>WHCV-R4 Reverse Complement
ACAACATTAACCTCCACACGCA
>WHCV-F5
GATCTCTCAAAGTGCCAGCTACAGT
>WHCV-R5 Reverse Complement
GGTGATGTGGTGGCTATTGATTATAA
>WHCV-F6
AGAAACTTTGTATTGCATAGACGGTG
>WHCV-R6 Reverse Complement
GGCATGCCTTCTTACTGTACTGGT
>WHCV-F7
GTTTAGCTGCTGTTAATAGTGTCCCTT
>WHCV-R7 Reverse Complement
GGTGATAGTGCGGAAGTTGCA
>WHCV-F8
TCCTACTGACCAGTCTTCTTACATCGT
>WHCV-R8 Reverse Complement
GTAGGCACGGCACTTGTGAAA
>WHCV-F9
GGTTTGCCTGGCACGATATTAC
>WHCV-R9 Reverse Complement
ATACAGCCAATCCTAAGACACCTAAGT
>WHCV-F10
TTGTCATCTCGCAAAGGCTCT
>WHCV-R10 Reverse Complement
TTTCCATGTGGGCTCTTATAATCTC
>WHCV-F11
GCTATGGGTATTATTGCTATGTCTGCT
>WHCV-R11 Reverse Complement
ATCAGCATTGTGGGAAATCCA
>WHCV-F12
CTGATCAAGCTATGACCCAAATGT
>WHCV-R12 Reverse Complement
GCAACAGCTGGACAATCCTTAAGT
>WHCV-F13
TCTGCGGTATGTGGAAAGGTTAT
>WHCV-R13 Reverse Complement
AGGAATTACTTGTGTATGCTGCTGAC
>WHCV-F14
AGGGCTTTAACTGCAGAGTCACAT
>WHCV-R14 Reverse Complement
AATTGCCGATAAGTATGTCCGC
>WHCV-F15
TCAATAGCCGCCACTAGAGGAG
>WHCV-R15 Reverse Complement
GTGACTGGACAAATGCTGGTGA
>WHCV-F16
TTGGGGCTTGTGTTCTTTGC
>WHCV-R16 Reverse Complement
ATTCCTTACACGTAACCCTGCTTG
>WHCV-F17
TGTCAATGCCAGATTACGTGCT
>WHCV-R17 Reverse Complement
GCTGTCCACGAGTGCTTTGTTA
>WHCV-F18
TATGGGCACATGGCTTTGAGT
>WHCV-R18 Reverse Complement
AAATGCCCGTAATGGTGTTCTTA
>WHCV-F19
TTGATGGACAACAGGGTGAAGTAC
>WHCV-R19 Reverse Complement
TTTATAAGCTCATGGGACACTTCG
>WHCV-F20
AGGAGTTGCACCAGGTACAGCT
>WHCV-R20 Reverse Complement
GTGCTGCAGCTTATTATGTGGGT
>WHCV-F21
CTATTAATTTAGTGCGTGATCTCCCTC
>WHCV-R21 Reverse Complement
TAACTCTATTGCCATACCCACAAATTT
>WHCV-F22
ACTTACTCCTACTTGGCGTGTTTATTC
>WHCV-R22 Reverse Complement
GGTGACATCTCTGGCATTAATGC
>WHCV-F23
CTATCATCTTATGTCCTTCCCTCAGTC
>WHCV-R23 Reverse Complement
TAGTCGTCGTCGGTTCATCATAAAT
>WHCV-F24
TACTTCAGGTGATGGCACAACAA
>WHCV-R24 Reverse Complement
AAGCTCACAAGTAGCGAGTGTTATCA
>WHCV-F25
CGTGTAGCAGGTGACTCAGGTTT
>WHCV-R25 Reverse Complement
GAATTCGTGGTGGTGACGGTA
>WHCV-F26
GGACCCCAAAATCAGCGAAAT
>WHCV-R26
AGTAGTGCTATCCCCATGTGATTTT
このfastaファイルを使ってBlast検索をします。
まずヒトゲノムで検索しました。
1番目のプライマにヒットしたヒトゲノムはこんな感じです。
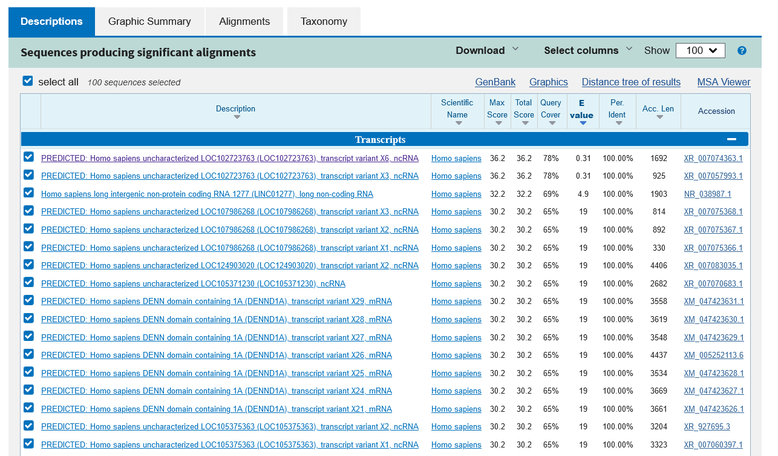
マッチする評価値が高い順に並んでいます。
最も評価値が高いゲノムの結果を見ると、100%の相同性となっていますが、下の図を見てもらうと完全一致しているわけではありません。
ここら辺が我々素人が騙されやすいところです。

1番目のプライマの長さは23塩基ですが、そのうち6番目から23番目の18塩基中、18塩基が一致しているから100%という意味だと分かります。
さて、この検索結果をテキストでダウンロードできます。
こんな感じです。
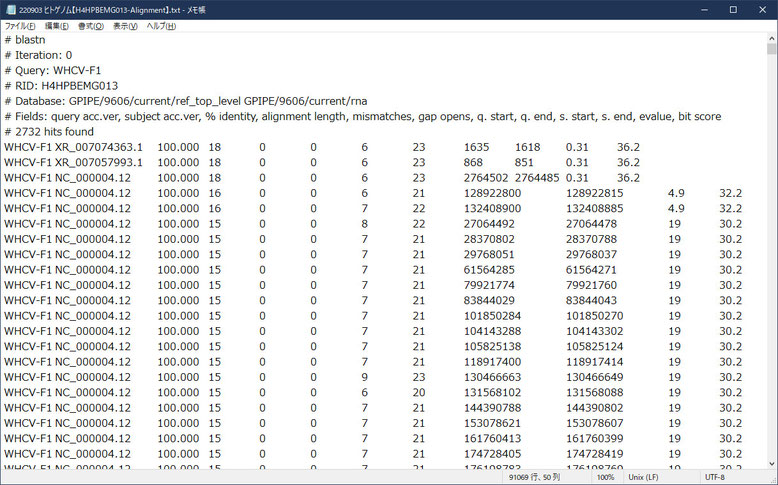
この結果から、複数のプライマに高いスコアでヒットしている第4染色体(NC_000004.12)について検証したいと思います。
手順は次の通りです。
- fastpで不良データを取り除いたSRR10971381をNC_000004.12にbowtie2-build、bowtie2、samtoolsを使ってマッピングします。
- マッピングできたリードデータを同様にして、MN908947.3にマッピングします。
- そしてsamtoolsを使ってカバレッジを求めます。
- MN908947.3にマッピングできたリードに対して、seqkitを用いて塩基長の分布を調べます。
まず、NC_000004.12にマッピングできたリードは次のようになりました。
| リードデータ(ペアエンドリード) | 本数 | 最短長 | 平均 | 最長長 |
| NC_000004.12にマッチしたリード(順方向+逆方向) | 512,300本 | 31塩基 | 119.1塩基 | 151塩基 |
このリードをMN908947.3にマッピングできたリードは次のようになりました。
| リードデータ(ペアエンドリード) | 本数 | 最短長 | 平均 | 最長長 |
| 更にMN908947.3にマッチしたリード(順方向+逆方向) | 242本 | 31塩基 | 34塩基 | 41塩基 |
このリードの塩基長の分布は下のようになりました。
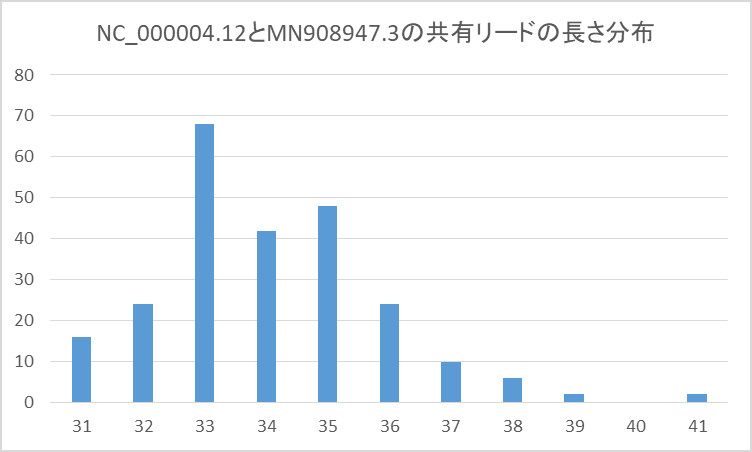
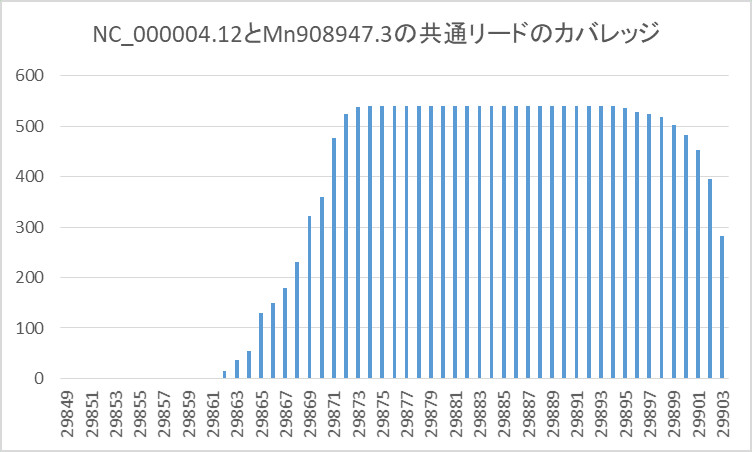
そして、カバレッジは下のようになりました。

青い線がカバレッジで、オレンジの線がプライマの位置です。
カバレッジがMN908947.3のお尻の部分に集中しています。
これでは分布がよく分からないので、お尻の部分を拡大した分布を示します。
まず、興味深いのが2つの塩基配列にマッチするリードの塩基長が31から41塩基という短いリードであること。
即ち、150塩基のリードが含まれていないこと。
そして、カバレッジがお尻の部分であること。
これは「33A」で有名な部分です。
アデニンが33個連続している部分を含んでいます。
多くの方はこれを見て新コロゲノムは恣意的に設計されたものだと主張しています。
そこの部分にヒトゲノムと共通するリードが存在すると言うのは興味深いです。
このデータだけでは何も分からないのが正直なところですが、一つ言えることは、「新コロゲノムはヒトゲノムが含まれている」ということが言えるということです。
これは重要な発見じゃないでしょうか。
他の染色体は?
新コロのプライマにヒットするヒト染色体は他にもあるので検証してみます。
ここでは、第1染色体(NC_000001.11)、第3染色体(NC_000003.12)、第8染色体(NC_000008.1)を調べてみます。
それぞれのゲノムとMN908947.3と共にマッチするリードは以下のようになりました。
| MN908947.3にマッチしたリードにマッチするゲノム | 本数 | 最短長 | 平均 | 最長長 |
| NC_000001.11 | 238本 | 31塩基 | 34塩基 | 41塩基 |
| NC_000003.12 | 242本 | 31塩基 | 34塩基 | 41塩基 |
| NC_000008.11 | 244本 | 31塩基 | 34塩基 | 41塩基 |
ほとんど同じような結果になりました。
リードの中身もほぼ同じでした。
ですから、リードの塩基長の分布もカバレッジもほぼ同じでした。
染色体の全てを調べたわけではないので結論付けることはできませんが、各ヒト染色体と共通する遺伝子配列と、新コロゲノムの遺伝子配列が共通する部分は、お尻の部分にあるということが分かりました。
これが何を意味するのかはまだ分かりません。
更に色々なゲノムで調べる必要があると思います。
リードの長さの意味
ヒト染色体と新コロゲノムと共にマッチするリードの特色として31~41塩基の短いリードであるということです。
恐らく150塩基の長さのリードはプライマで大量にコピーされたリードですから、この短いリードはプライマに関係なかったリードだと予想されます。
リードの長さ分布で本数が少ない短いリードは自然界に存在する遺伝物質由来の遺伝子配列なのかもしれません。
Blast検索で新コロプライマにヒットするヒトゲノムのリストを見て分かるように、プライマが完全一致する部分はありませんでした。
これはバクテリアでも一緒です。
新コロゲノムが創作物という前提に立った時、150塩基の長さに渡って一致する塩基配列が自然界に存在する遺伝子物質には無いのかもしれません。
だとすると、どうやってこの150塩基のリードが作られたのか?という疑問が湧いてきます。
しかも、感染研は再現したと主張しているわけですから、武漢以外で自然界に存在しない遺伝子配列を持つリードをどうやって用意させるのか?という疑問も湧いてきます。
ちょっと考えると、自然界に存在する遺伝子配列から簡単に150塩基のリードが見つかってしまえば、この新コロゲノムはキメラ配列であることがバレてしまうので、物事はもっと巧妙なのかもしれません。
ウイルスの分離における仮想化技術は、細胞培養において本来はエラーになる御法度を使っていました。
つまり、栄養の欠乏の状態を作っていました。
この遺伝子配列再現においても、本来であればエラーになる方法を使っているのかもしれません。
それはPCR技術の中にあるのかもしれません。



