2-4節
公開リードデータの謎
前々節と前節で紹介した「W+」の論文ですが、その続編が掲載されました。
- einem Mathematiker aus Hamburg:
「Strukturelle Analyse von Sequenzdaten in der Virologie」,
WISSENSCHAFFTPLUS magazin, 1/2022.
この論文の内容は難しくて私の理解力では全てを把握することはまだまだ時間がかかりそうですが、本節では私が理解した部分を私になりに解釈した表現で説明したいと思います。
この分野に詳しい方は是非、原著に触れて欲しいと思います。
因みにこれまで著者名を紹介していませんでした。
ドイツ語が分かる方なら気付いていらっしゃると思いますが、著者名が書かれていないのです。
「ハンブルグ出身の数学者で、まだ無名であることを望んでいる」と書かれています。
なんかお茶目です。
不思議なリードの長さ
まずこの図を見ていただきたい。
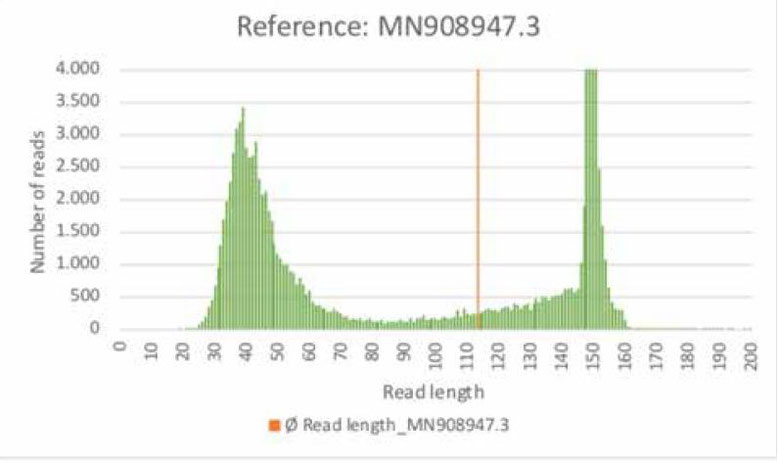
これは新型コロナウイルスの遺伝子配列(アクセッション番号:MN908947.3)をリファレンス配列として、公開リードデータ(SRR10971381)をマッピングした時に、マップすることができたリードを取り出して、リードの長さとその数をグラフにしたものです。
明らかにおかしなグラフが得られています。
ランダムに遺伝子物質を切ったのであれば、その長さは正規分布に従うはずです。
ですが、得られたグラフは約20塩基から80塩基にかけて正規分布のような形をしている領域と、140塩基以降の搭のような形、まるで揃えて切ったような印象を持つ分布に分かれています。
明らかに不自然です。
これのカバレッジが前節でお見せしたグラフです。
再掲します。

このグラフのデータを下に示します。
| リファレンス配列 | MN908947.3 |
| ゲノムの長さ | 29,903塩基 |
| マッチしたリードの数 | 121,779本 |
| 平均リード長 | 145.56塩基 |
| カバーした塩基の数 | 29,903塩基 |
| カバー率 | 100.00% |
次に100塩基以上を除外して、新型コロナウイルスの遺伝子配列にマッピングすると、次のようなカバレッジのグラフが得られました。
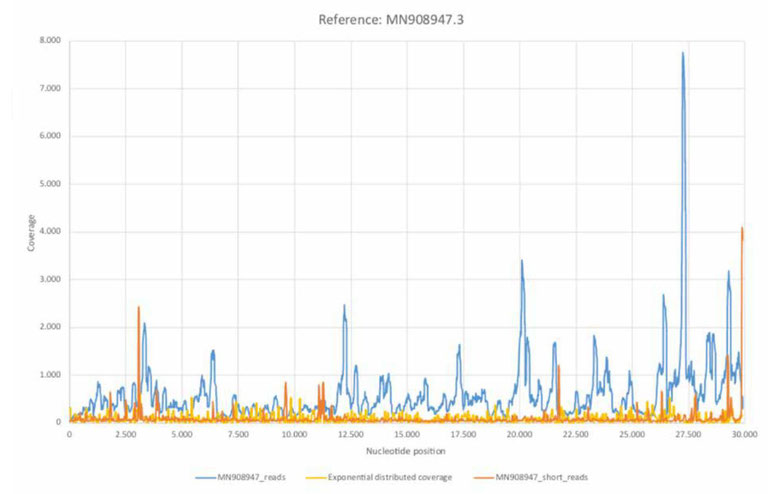
赤い線が100塩基以下のリードでマッピングしたカバレッジで青い線が全てのリードでマッピングしたカバレッジです。
このグラフのデータは以下の通りです。
| リファレンス配列 | MN908947.3 |
| ゲノムの長さ | 29,903塩基 |
| マッチしたリードの数(100塩基長未満) | 59,949本 |
| 平均リード長 | 46.24塩基 |
| カバーした塩基の数 | 29,903塩基 |
| カバー率 | 100.00% |
赤い線と青い線は、分布の仕方がまるで異なります。
赤い線はプライマの位置で極端に多いわけでもないことが分かります。
つまり、先ほどのグラフで、自然な山に分布している比較的短いリードは、新型コロナウイルスゲノム配列を偏りなく均等にマッピングされるということです。
逆に、150塩基長付近の人工的なリードはプライマ付近に偏って分布しているということが分かります。
これらの人工的リードは、最初から存在していたのか、それとも公開のリードに後から追加されたのでしょうか。
HIVをリファレンス配列にして比較する
論文では他のウイルスの遺伝子配列でも比較検討しています。
まずHIV(アクセッション番号:LC312715.1)で検討しています。
なぜこの遺伝子配列を選択したのかは、論文を何度読んでも分からなかったのですが、公開されたリードデータと高い構造類似性が見られたからと書いてあります。
カバレッジを示すグラフは下図のようになりました。

このグラフの青い線がカバレッジ、オレンジの線がプライマの位置です。
この結果の様々なデータは以下の通りです。
| リファレンス配列 | LC312715.1 |
| ゲノムの長さ | 8,819塩基 |
| マッチしたリードの数 | 65,196本 |
| 平均リード長 | 51.84塩基 |
| カバーした塩基の数 | 8,819塩基 |
| カバー率 | 100.00% |
リファレンス配列にマッピングされたリードの長さの分布は以下の通りです。
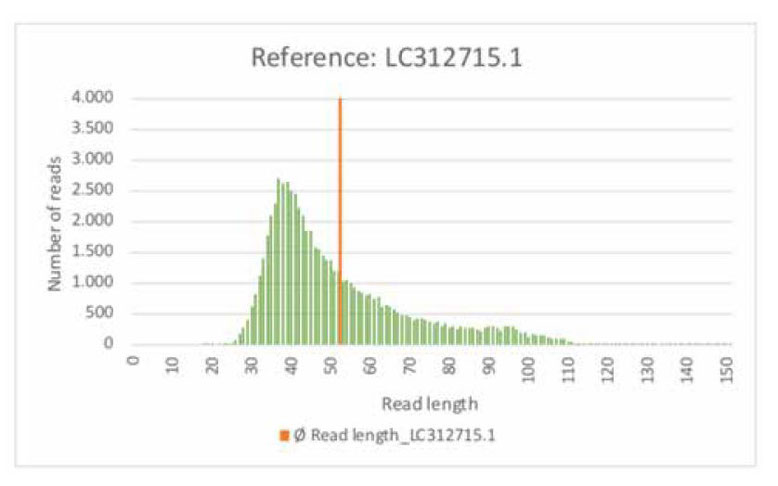
カバレッジのグラフを見るとプライマの付近で高いカバレッジが見られます。
ですが、新型コロナウイルスのような150塩基長のリードはほとんど使われていません。
つまり、プライマで増幅された短いリードしかマッピングされなかったということです。
150塩基長の長いリードはHIVウイルスの構造には無関係だったということが言えます。
可能な方は是非論文で確認していただきたいのですが、他のリファレンス配列でも検討していますが、150塩基長のリードは新型コロナウイルスのゲノム配列しかマッピングされていませんでした。
他のリファレンス配列では最初に掲載したグラフのような2つの分布を示すことはなく、このHIVと同じような1つの正規分布的な分布を示しています。
このことから、プライマで増幅された長いリードは、新型コロナウイルスの構造に大きく関与していると言えます。
言い換えれば、150塩基長の長いリードは、アセンブルして新型コロナウイルスゲノム配列を再現するのに重要なリードであると考えられます。
特異的プライマとPCRで遺伝子配列を再現する
前節で新型コロナウイルスのゲノム配列を再現するにはアセンブラの選択が重要であると主張した論文を紹介しました。
この主張とは異なり、新型コロナウイルスに特異的なプライマとPCRで検体を増幅することでゲノム配列を再現することができるという論文があります。
- Clinton R. Paden et al.:
「Rapid, Sensitive, Full-Genome Sequencing of Severe Acute Respiratory Syndrome Coronavirus 2」,
Emerg Infect Dis. 2020 Oct; 26(10): 2401–2405.
この論文はA459ヒト細胞株の全核酸を含んだサンプルを用いて、設計したプライマでPCR増幅させ、リードが新型コロナウイルスのゲノム配列の全域を覆うことができるかを調べています。
Ct値が35までが効果的であると言っています。
-
Annika Brinkmann et al.:
「AmpliCoV: Rapid Whole-Genome Sequencing Using Multiplex PCR Amplification and Real-Time Oxford Nanopore MinION Sequencing Enables Rapid Variant Identification of SARS-CoV-2」,
Front Microbiol. 2021; 12: 651151.
上記論文と同様に検体をPCR増幅して、リードが新型コロナウイルスのゲノム配列を覆うことができるかを調べています。Ct値が大きいとシーケンスできなかったが、Ct値が35であれば可能だったと言っています。
正直、これらの論文の内容を正確に理解しているわけではありませんが、新型コロナウイルスに特異的なプライマを使用することで、新型コロナウイルスのゲノム配列を再現できるリードデータを作成することが可能であることを示唆しているものと思われます。
まとめ
- 公開されたリードデータには不自然な人工的に切りそろえたと思われる長さのリードが含まれている。
これらのリードはプライマの位置に偏って分布している。
このことから、プライマの設計が新型コロナウイルスの再現性に大きく関わっていると思われる。
- PCRのCt値の選択も、新型コロナウイルスのゲノム配列の再現性に関係があると思われる。
高いCt値(35)によるPCR増幅によって、検体には含まれていなかった遺伝子配列が生じた可能性も考慮する必要がある。
この可能性を考慮して対照実験を行うべきである。
- 中国論文で最初に使用したリードデータを公開するべきである。
ジキ仮説
今回紹介した2つの論文から、次のことを考えました。
- 中国論文に書かれている処理は実際に行われている。
- その結果得られたリードデータに対してMegahitとTriniryでアセンブルした。
アセンブルした結果得られたコンティグも論文の記載通りだった。
- 中国論文に記載されている補足表を見ると得られたコンティグはヒト細胞と一致している者が見られないので、検体からヒト由来の遺伝子物質が除去されているのも本当だろう。
- Megahitで得られた最長コンティグ(30,474塩基)には、ヒト由来の遺伝子配列が含まれていないため、欲しい遺伝子配列の形になっていなかったため、配列アライメントをした
- この最長コンティグを再現することができないので、コウモリコロナウイルスSL-CoVZC45にどれだけ一致しているのかは分からない。
全く一致していない可能性も考えられる。
- この最長コンティグにヒト由来の遺伝子配列が含まれていない可能性から考えると、この最長コンティグから新型コロナウイルスのゲノム配列が本当に得られたのか疑わしい。
全く別物の可能性も考えられる。 - 新型コロナウイルスのゲノム配列(29,903塩基)は最初から用意されていた。
コロナウイルス属のヒトコロナウイルスSARS-CoV Tor2と コウモリコロナウイルスSL-CoVZC45をベースに設計されたかもしれない。
- 新型コロウイルスのゲノム配列からプライマを設計した。
- 患者からの検体から抽出したRNAから得られるcDNAに対して、設計したプライマを用いてPCR増殖した(Ct値35)。
この処理で以て150塩基長のリードを大量にコピーした。 - 以上の処理で得られたサンプルを次世代シーケンサーで読み取り、リードデータを作った。
そのリードデータでアセンブルすると、新型コロナウイルスのゲノム配列を再現できることを確認した。
- そのリードデータを公開した。
- ヒト由来の遺伝子物質が含んだ検体を中国論文で設定されたプライマとCt値35のPCRによって特定の遺伝子物質を増幅してやれば、それから得られるリードデータをアセンブルすると新型コロナウイルスのゲノム配列を再現できる可能性がある。
これはあくまでも仮説です。
ですが、ステファン・ランカ氏が行った対照実験で、ゲノム配列の再現も行っていると聞いているので、その結果が公表されたときに、遺伝子配列の謎が全て解明されると思われます。





